Coronavirus, la ricerca della cura passa anche dal tuo PC
Si pensa che il ripiegamento proteico sia la chiave per trovare una cura.
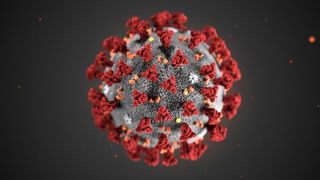
Il Coronavirus è ormai un’emergenza sanitaria pubblica di interesse internazionale. A questo proposito, ci si domanda se possa esserci un modo per aiutare gli scienziati a velocizzare la ricerca di una cura.
Nel 2000, alcuni ricercatori di Stanford hanno ideato un programma chiamato Folding@home, che intende sfruttare la potenza dei PC di qualsiasi utente medio per svolgere calcoli e generare dati in grado di permettere ai ricercatori di studiare più rapidamente il virus. L’obiettivo è quello di riuscire a neutralizzare il coronavirus nelle prime fasi dell’infezione.
Folding@home (FAH) è un progetto che utilizza il calcolo distribuito per simulare e studiare diversi fenomeni come il ripiegamento delle proteine, la progettazione di farmaci e altri tipi di dinamiche molecolari.
Naturalmente, in questo momento gli sviluppatori sono focalizzati nel rendere il progetto compatibile con la ricerca di una cura definitiva per il Covid-19. La Stanford University ha comunicato che chiunque può parteciparvi, mettendo a disposizione il proprio computer connesso a Internet per eseguire simulazioni utili al team di ricercatori.
Cosa si intende per ripiegamento proteico?
Prima di inoltrarci nell’argomento, cerchiamo di capire come funziona la struttura delle proteine. L’enciclopedia Treccani riporta la seguente definizione di “ripiegamento delle proteine”, definendolo un “processo biochimico con il quale un polipeptide con una struttura casuale si ripiega spontaneamente nell’unica struttura tridimensionale possibile dotata di attività biologica.
L’informazione necessaria per ottenere il ripiegamento (folding) di una proteina nella struttura tridimensionale attiva, detta conformazione nativa, è interamente contenuta nella struttura primaria della proteina stessa.
Il fenomeno del ripiegamento delle proteine è influenzato dalla composizione del mezzo nel quale queste si trovano disciolte e, nella maggior parte dei casi, si tratta di un processo che avviene nelle cellule al termine o, a volte, addirittura durante la sintesi delle proteine stesse.
Get the best Black Friday deals direct to your inbox, plus news, reviews, and more.
Sign up to be the first to know about unmissable Black Friday deals on top tech, plus get all your favorite TechRadar content.
Sembra sempre più probabile che le molecole proteiche siano aiutate in vivo a raggiungere la conformazione tridimensionale definitiva da un gruppo di particolari proteine, dette chaperonine o chaperon molecolari, che guidano le varie fasi del ripiegamento.”
In altre parole, per ripiegamento delle proteine si intende il meccanismo attraverso il quale una proteina assume una conformazione finale stabile e funzionale. Si tratta di un processo necessario affinché una proteina possa passare dalla sua struttura primaria a quella effettivamente funzionale.
Le cellule bersaglio primarie, attraverso le quali il Covid-10 è in grado di moltiplicarsi, sono quelle epiteliali del tratto respiratorio e gastrointestinale. Come tutti i virus, anche questo muta in continuazione, modificando il suo aspetto per essere in equilibrio con il sistema immunitario ospite.
A trasformarsi è la proteina di superficie chiamata 'spike', che si aggancia alle cellule umane, quindi subisce un cambiamento strutturale che consente alla membrana virale di fondersi con la membrana cellulare. Tra i meccanismi mirati a fermare l’azione del virus, vi è lo sviluppo di molecole che inibiscono l’interazione della proteina spike.
- Scoprite Disney+, intrattenimento senza fine per tutta la famiglia
- iPhone 12: data di uscita, prezzo, notizie
- iPhone 9: data d’uscita, prezzo, notizie e indiscrezioni
Per l’elaborazione di nuovi farmaci e di strategie di attacco al virus, ci viene incontro la tecnologia, che ci permette di contribuire direttamente alla ricerca. Il suddetto procedimento intende sfruttare la potenza di calcolo dei computer messi a disposizione da chiunque in tutto il mondo.
Gli utenti che vi aderiscono rendono accessibile la potenza di calcolo dei propri computer i quali - quando non hanno altre operazioni in coda - eseguono calcoli matematici per conto dei ricercatori, e inviano loro i risultati. I calcoli assegnati rappresentano tasselli di complesse simulazioni pensate per prevedere il comportamento di determinate proteine.
Guida all’installazione e all’uso di Folding@Home
- Scaricate e installate Folding@Home come fareste con un comune programma.
- Utilizzate le opzioni di installazione personalizzata per impostare le preferenze di avvio desiderate.
- Eseguite Folding@Home. Verrete reindirizzati automaticamente al controllo web.
- [Opzionale] Inserite un nome e unitevi al nostro team: 40051
Dopo aver completato questi brevi e semplici passaggi, il sistema sarà pronto per eseguire automaticamente le simulazioni di ripiegamento delle proteine. Gli utenti potranno regolare alcune impostazioni per stabilire quanta potenza di elaborazione mettere a disposizione dalla propria CPU e dalla scheda video.
A causa dell'allocazione delle risorse rimanenti sul PC consumerete più energia e, di conseguenza, la vostra bolletta sarà più salata, ma ne varrà la pena dato che si tratta di una buona causa.
Via Tomshardware
